自閉スペクトラム症の多発罹患家系において,稀な変異が発症に重要な役割を果たすことが示唆されている.自閉スペクトラム症の稀なリスク変異を同定するため,われわれは4人の罹患者(同胞2人,母方いとこ2人)を有する多発罹患家系の全エクソームシークエンスおよび症例・対照サンプル(243対667)を用いたフォローアップ研究を実施した.多発罹患家系の4人(発端者,罹患同胞,非罹患同胞,保因者と推定される母)について全エクソームシークエンスを行ったところ,2つの稀な短縮型変異(RPS24遺伝子Q191X変異とCD300LF遺伝子P261fsX266変異)を同定した.これらの変異は,フォローアップ研究の910サンプルでは検出されなかった.本研究により,2つの稀な短縮型変異(RPS24遺伝子Q191X変異とCD300LF遺伝子P261fsX266変異)が自閉スペクトラム症の候補リスク変異であることが示唆された.
2)新潟大学医歯学総合病院魚沼地域医療教育センター精神科
3)新潟大学大学院医歯学総合研究科「システム脳病態学の確立とそれを用いた臨床研究推進事業」担当
4)新潟大学大学院医歯学総合研究科地域精神医療学寄附講座
5)大島病院
6)新潟大学医学部医学科総合医学教育センター
はじめに
自閉スペクトラム症(autism spectrum disorder:ASD)は,社会的コミュニケーションの欠陥,限定され反復的な行動や興味の様式などの特徴をもつ神経発達障害である.その病態は不明であるが,遺伝要因と環境要因が相互に影響して発症に至る多因子疾患であり,遺伝率は52.4%と見積もられ,その大部分は頻度の高いリスク多型に起因すると推定されている9).しかし,個々のリスク多型がASDの発症に与える影響は小さいことから,ASDの病態解明につながる分子基盤を得るには,頻度は稀だが相対危険度の高い変異を同定することが重要と考えられる.
近年では高速シークエンサーの普及に伴い,ゲノム中の全エクソンを網羅的にシークエンスする全エクソームシークエンス(whole-exome sequencing:WES)が実施され,ASDの発症に大きな影響をもつリスク変異を効率的に同定できるようになった.発端者・両親トリオのWES研究では,稀なde novo変異がASDの発症に重要な役割を果たすことが示された13)17)18)20).ASDの発症に大きな影響を与えるリスク変異が存在する場合には,家系内に複数の罹患者が認められる可能性が高いと考えられるため,多発罹患家系を用いたWES研究を行うことも重要である.しかしASD多発罹患家系のWES研究はほとんど実施されていない22)24).血族婚家系では,ホモ接合体および複合ヘテロ接合体変異がASDと共分離していた24).ASD多発罹患10家系では,両親から罹患同胞に伝達されたヘテロ接合体短縮型変異の数は,伝達されなかったものよりも有意に多かったと報告されている22).これらのことから,ASD多発罹患家系では両親から伝達された稀な変異がASDの発症に関与することが示唆されているが,結論を得るにはさらなる研究が必要である.
そこでわれわれは,ASD多発罹患家系のWES研究を実施するとともに,症例・対照サンプルを用いたフォローアップ研究を行った.
I.研究の方法および結果
1.対象および方法
本研究は新潟大学医学部遺伝子倫理審査委員会で承認されており,全ての対象者およびその家族から書面にて研究参加の同意を得た.対象者は全て日本人である.
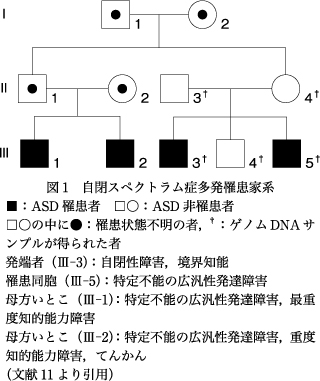
ASD多発罹患家系(図1)には4人の罹患者がいる.自閉性障害と境界知能をもつ発端者(III-3),特定不能の広汎性発達障害をもつ同胞(III-5),特定不能の広汎性発達障害と最重度知的能力障害をもつ母方いとこ(III-1),特定不能の広汎性発達障害,重度知的能力障害,てんかんをもつ母方いとこ(III-2)である.罹患者の診断は,精神疾患の診断・統計マニュアル第4版(Diagnostic and Statistical Manual of Mental Disorders, Fourth Edition:DSM-IV)に従って,児童精神科医により患者や家族の非構造化面接などに基づいてなされた.発端者の両親(II-3,II-4)は児童精神科医の非構造化面接により非罹患者と確認された.他の家族(I-1,I-2,II-1,II-2)は面接できなかったため罹患状態を不明とした.
フォローアップ研究の対象者はASD患者243人(男性191人,女性52人,平均年齢18.1±8.6歳)と対照667人(男性341人,女性326人,平均年齢38.3±10.8歳)からなる.患者群の診断内訳は,自閉性障害72人,アスペルガー障害107人,特定不能の広汎性発達障害64人である.
多発罹患家系のなかでは,発端者(III-3),非罹患同胞(III-4),罹患同胞(III-5),非罹患両親(II-3,II-4)のDNAサンプルが得られた.発端者(III-3),非罹患同胞(III-4),罹患同胞(III-5),保因者と推定される母(II-4)についてWESを実施した.SureSelect Human All Exon V5 Kit(アジレント社)を用いて全エクソン領域を濃縮し,HiSeq2000(イルミナ社)によりシークエンスを行った.シークエンスリードは,Burrows-Wheeler Aligner(http://biobwa.sourceforge.net/)を用いて参照ヒトゲノム(UCSC19)にマッピングされた.PCR産物の重複はPicard(http://picard.sourceforge.net/)を用いて取り除かれた.変異はGenome Analysis Toolkit(http://www.broadinstitute.org/gatk/)を用いて検出され,SnpEff(http://snpeff.sourceforge.net/)により注釈づけされた.
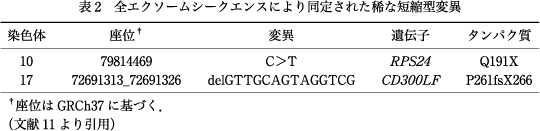
変異を絞り込むため,次のフィルタリング手順を適用した(表1).①当該塩基をカバーするシークエンスリードの数が10未満の変異を除外する.②発端者(III-3)と罹患同胞(III-5)が共有する.③非罹患同胞(III-4)は保有しない.④保因者と推定される母(II-4)から伝達された可能性のある変異を含める.⑤SnpEffを用いて高影響と予測された変異を含める.⑥Human Genetic Variation Database(http://www.genome.med.kyotou.ac.jp/SnpDB/)またはEnsembl Genome Browser(http://asia.ensembl.org/index.html)上の1000人ゲノムプロジェクトの日本人データにおいて変異アレル頻度が0.01未満の変異を選ぶ.以上の手順により絞り込まれた2つの短縮型変異(RPS24遺伝子Q191X変異とCD300LF遺伝子P261fsX266変異)について,サンガーシークエンスにより存在を確認した.
次に,多発罹患家系のWESで同定された稀な変異とASDとの関連を調べるため,症例・対照サンプル(243対667)を用いたフォローアップ研究を行った.RPS24遺伝子Q191X変異はTaqMan法(アプライドバイオシステムズ社),CD300LF遺伝子P261fsX266変異はフラグメント解析を用いてタイピングされた.
2.結果
多発罹患家系の4人のWESでは合計で202,401個の変異が検出された(表1).フィルタリング手順を適用し,われわれは2つの稀な短縮型変異を同定した(表2).1つはRPS24遺伝子の一塩基変異(chr10:g.79814469C>T)で,本来はグルタミン(Q)を指定する191番目のコドンが終止コドン(X)となる.もう1つは,CD300LF遺伝子の14塩基欠失(chr17:g.72691313_72691326delGTTGCAGTAGGTCG)で,本来はプロリン(P)を指定する261番目のコドンからフレームシフト(fs)を起こし266番目のコドンが終止コドン(X)となる.これらの変異はサンガーシークエンスにより確認された.
次に,われわれは患者243人と対照667人についてRPS24遺伝子Q191X変異とCD300LF遺伝子P261fsX266変異をタイピングしたが,910人全員が変異アレルを保有していなかった.
II.考察
1.考察
本研究では,ASD多発罹患家系のWESによって2つの稀な短縮型変異(RPS24遺伝子Q191X変異とCD300LF遺伝子P261fsX266変異)が同定されたが,フォローアップ研究の910人ではこれらの変異は検出されなかった.RPS24遺伝子Q191X変異とCD300LF遺伝子P261fsX266変異は多発罹患家系のみに同定されたことから,これらの変異が特定の家系ではASDの発症に重要な役割を果たす可能性が示唆された.
RPS24遺伝子は,リボソーム新生に関与するリボソームタンパク24(RPS24)をコードしている3).別のリボソームタンパク遺伝子であるRPL10遺伝子の稀なミスセンス変異(L206M変異とH213Q変異)もまた,ASD罹患同胞を有する2つの異なる家系で同定されている14).また,これらの変異はリボソームサブユニットの生成に変化をきたすことも明らかにされている14).したがって,リボソーム新生の機能不全がASD発症に関与するものと考えられる.RPS24遺伝子はDiamond-Blackfan貧血3(DBA3)の原因遺伝子であり,患者では以下の4つのヘテロ接合変異が報告されている10)15).すなわち,Q106X変異(rs104894188),R16X変異(rs104894189),エクソン2のスキップを生じるイントロン1とエクソン2境界の欠失・挿入(rs116840806),エクソン1・2およびエクソン3の一部を含む欠失である.ASD多発罹患家系において,発端者,罹患同胞,母親はQ191X変異のヘテロ接合体であるが,DBA3の特徴的症候(貧血,骨髄赤芽球減少症,先天奇形など)は認めなかった.このことから,Q191X変異はDBA3の原因変異ではないと考えられる.RPS24遺伝子のmRNA発現レベルは,胎児脳で高く成人脳では低い10).この胎児優位な発現パターンは,症候性神経発達障害,知的能力障害,ASDに関連する遺伝子群の発現パターンと一致する1).以上のことから,RPS24遺伝子Q191X変異が,ASDの基底をなすある種の神経発達機能障害を引き起こしうると推測される.しかしその機能的意義は依然解明されていない.
CD300LF遺伝子はCD300遺伝子複合体の1つであり,CMRF35様分子1(CLM-1)をコードし,CD300fとも呼ばれる2).CLM-1は骨髄細胞に発現する免疫受容体であり,Cd300lf遺伝子ノックアウトマウスは,重篤な自己免疫性脳脊髄炎を生じる23).しかし,CD300LF遺伝子P261fsX266変異保有者において脳脊髄炎の既往は認めなかった.CLM-1の過剰発現は,ラットの線条体と大脳皮質へのN-methyl-D-aspartate注入による急性脳障害を軽減した19).このことから,CLM-1は神経炎症に対して保護的作用を有するといえる.脳内の慢性炎症がASDの病態に関与することを示唆する報告もある6).これらの知見から,CD300LF遺伝子P261fsX266変異が慢性神経炎症を引き起こす結果として,ASD発症に至るのかもしれない.
2.苦労・工夫したこと
ASDのWES研究に関して日本は後れをとっているが,ASD発症に大きな影響をもつ稀なリスク変異を同定できるよう,本研究では優れた研究デザインを組むように工夫した.多発罹患家系のWES研究は疾患発症リスクに大きな影響をもつ稀な変異の同定に有用と考えられている4).そのため,本研究では4人のASD罹患者を有する家系サンプルを収集しWESを実施した.ただし,罹患者である母方いとこ2人のゲノムDNAサンプルを得られず,彼らがRPS24遺伝子Q191X変異とCD300LF遺伝子P261fsX266変異を保有しているか否かを確認できなかった.また,多発罹患家系のWESにより同定された候補リスク変異と疾患との関連を,独立サンプルを用いて確認することも重要である5).そこで,症例・対照サンプル(243対667)を用いたフォローアップ研究により,RPS24遺伝子Q191X変異とCD300LF遺伝子P261fsX266変異が特定の家系のみでなく一般にASDの発症リスクに大きな影響を有していることの確認を試みた.これらの変異は計910サンプルでは同定されなかったが,変異アレル頻度が極めて低く,フォローアップ研究のサンプルサイズが十分ではないために検出できなかった可能性がある.
3.意義
本研究ではASD発症に大きな影響を有する可能性のある2つの稀な短縮型変異(RPS24遺伝子Q191X変異とCD300LF遺伝子P261fsX266変異)を見出した.
おわりに―今後の課題および方向性―
われわれはASD罹患者が複数いる他の家系についてもWES研究を進めている.ASD罹患状態一致一卵性双生児を有する1家系のWES研究では,3つの稀な短縮型変異(PCDHA9遺伝子L840fs変異,WDR90遺伝子V1125fs変異,EFCAB5遺伝子L1210fs変異)が同定された7).このうちWDR90遺伝子V1125fs変異とEFCAB5遺伝子L1210fs変異は症例・対照サンプル(257対667)でタイピング可能であったが,変異アレルは検出されなかった.また,ASD罹患同胞3人を有する2家系のWES研究では,6つの稀なミスセンス変異が同定され,そのうちCLN8遺伝子R24H変異は1つの家系で罹患者である父から罹患同胞3人へ伝達していた8).さらにASD罹患者256人でCLN8遺伝子コード領域をリシークエンスしたところ,5つの稀なミスセンス変異を同定した(R24H変異,F39 L変異,R97H変異,T108M変異,N152S変異)12).しかし,症例・対照サンプル(568対1,017)を用いた関連解析ではいずれの変異もASDとの有意な関連を認めなかった.サンプルサイズが十分とはいえないことから,より多くの施設との共同研究を行うことで,検出力の向上をはかる必要がある.
ASDの発症に大きな影響を及ぼすリスク変異を同定した後には,機能解析を行い発症機序解明に結びつけることが重要である.ヒト多能性幹細胞16)や変異ノックインマウスを用いたモデル研究21)などへと展開することで,ASDの発症機序を解明し,さらには新たな診断・治療法の開発や療育のオーダーメイド化につなげることが最終目標と考えている.
本論文は,PCN誌に掲載された最新の研究論文11)を編集委員会の依頼により,著者の1人が日本語で書き改め,その意義と展望などにつき加筆したものである.
本研究は科学研究費補助金(No. 26860917)の助成を受けて行われた.
なお,本論文に関連して開示すべき利益相反はない.
謝 辞 研究に参加いただいたすべての方々に謝意を表します.
1) Birnbaum, R., Jaffe, A. E., Hyde, T. M., et al.: Prenatal expression patterns of genes associated with neuropsychiatric disorders. Am J Psychiatry, 171; 758-767, 2014![]()
2) Borrego, F.: The CD300 molecules: An emerging family of regulators of the immune system. Blood, 121; 1951-1960, 2013![]()
3) Choesmel, V., Fribourg, S., Aguissa-Touré, A. H., et al.: Mutation of ribosomal protein RPS24 in Diamond-Blackfan anemia results in a ribosome biogenesis disorder. Hum Mol Genet, 17; 1253-1263, 2008![]()
4) Cirulli, E. T., Goldstein, D. B.: Uncovering the roles of rare variants in common disease through whole-genome sequencing. Nat Rev Genet, 11; 415-425, 2010![]()
5) Cruchaga, C., Karch, C. M., Jin, S. C., et al.: Rare coding variants in the phospholipase D3 gene confer risk for Alzheimer's disease. Nature, 505; 550-554, 2014![]()
6) Depino, A. M.: Peripheral and central inflammation in autism spectrum disorders. Mol Cell Neurosci, 53; 69-76, 2013![]()
7) Egawa, J., Watanabe, Y., Sugimoto, A., et al.: Whole-exome sequencing in a family with a monozygotic twin pair concordant for autism spectrum disorder and a follow-up study. Psychiatry Res, 229; 599-601, 2015![]()
8) Egawa, J., Watanabe, Y., Wang, C., et al.: Novel rare missense variations and risk of autism spectrum disorder: whole-exome sequencing in two families with affected siblings and a two-stage follow-up study in a Japanese population. PLoS One, 10; e0119413, 2015![]()
9) Gaugler, T., Klei, L., Sanders, S. J., et al.: Most genetic risk for autism resides with common variation. Nat Genet, 46; 881-885, 2014![]()
10) Gazda, H. T., Grabowska, A., Merida-Long, L. B., et al.: Ribosomal protein S24 gene is mutated in Diamond-Blackfan anemia. Am J Hum Genet, 79; 1110-1118, 2006![]()
11) Inoue, E., Watanabe, Y., Egawa, J., et al.: Rare heterozygous truncating variations and risk of autism spectrum disorder: Whole-exome sequencing of a multiplex family and follow-up study in a Japanese population. Psychiatry Clin Neurosci, 69; 472-476, 2015![]()
12) Inoue, E., Watanabe, Y., Xing, J., et al.: Resequencing and association analysis of CLN8 with autism spectrum disorder in a japanese population. PLoS One, 10; e0144624, 2015![]()
13) Iossifov, I., Ronemus, M., Levy, D., et al.: De novo gene disruptions in children on the autistic spectrum. Neuron, 74; 285-299, 2012![]()
14) Klauck, S. M., Felder, B., Kolb-Kokocinski, A., et al.: Mutations in the ribosomal protein gene RPL10 suggest a novel modulating disease mechanism for autism. Mol Psychiatry, 11; 1073-1084, 2006![]()
15) Landowski, M., O'Donohue, M. F., Buros, C., et al.: Novel deletion of RPL15 identified by array-comparative genomic hybridization in Diamond-Blackfan anemia. Hum Genet, 132; 1265-1274, 2013![]()
16) Mariani, J., Coppola, G., Zhang, P., et al.: FOXG1-dependent dysregulation of GABA/Glutamate neuron differentiation in autism spectrum disorders. Cell, 162; 375-390, 2015![]()
17) Neale, B. M., Kou, Y., Liu, L., et al.: Patterns and rates of exonic de novo mutations in autism spectrum disorders. Nature, 485; 242-245, 2012![]()
18) O'Roak, B. J., Vives, L., Girirajan, S., et al.: Sporadic autism exomes reveal a highly interconnected protein network of de novo mutations. Nature, 485; 246-250, 2012![]()
19) Peluffo, H., AlíRuiz, D., EjarqueOrtíz, A., et al.: Overexpression of the immunoreceptor CD300f has a neuroprotective role in a model of acute brain injury. Brain Pathol, 22; 318-328, 2012![]()
20) Sanders, S. J., Murtha, M. T., Gupta, A. R., et al.: De novo mutations revealed by whole-exome sequencing are strongly associated with autism. Nature, 485; 237-241, 2012![]()
21) Tabuchi, K., Blundell, J., Etherton, M. R., et al.: A neuroligin-3 mutation implicated in autism increases inhibitory synaptic transmission in mice. Science, 318; 71-76, 2007![]()
22) Toma, C., Torrico, B., Hervás, A., et al.: Exome sequencing in multiplex autism families suggests a major role for heterozygous truncating mutations. Mol Psychiatry, 19; 784-790, 2014![]()
23) Xi, H., Katschke, K. J. Jr., Helmy, K. Y., et al.: Negative regulation of autoimmune demyelination by the inhibitory receptor CLM-1. J Exp Med, 207; 7-16, 2010![]()
24) Yu, T. W., Chahrour, M. H., Coulter, M. E., et al.: Using whole-exome sequencing to identify inherited causes of autism. Neuron, 77; 259-273, 2013![]()