統合失調症(SZ)と双極性障害(BD)は遺伝要因と環境要因を共有することが知られており,複雑な遺伝環境相互作用が発症に関与すると考えられている.ゲノムワイド関連解析の結果,主要な精神疾患のなかでもSZとBDは遺伝要因の重なりが特に大きいことが知られているが,エピゲノム要因の重なりについては十分な検討がなされていない.本研究では,SZで行われた大規模なゲノムワイドメチル化関連解析(MWAS)で同定された上位5ヵ所の候補領域について,BD試料を用いた検討を行った.5ヵ所の候補領域のうち2ヵ所〔FAM63B,染色体16番intergenic領域(IR in chr 16)〕では,SZと同様に,BDにおいても有意な低メチル化状態が認められ,SZとBDに共通なエピゲノム要因である可能性が示唆された.また,1ヵ所(TBC1D22A)の領域では,SZとは異なり,BDでは健常者に比して有意な高メチル化状態が認められた.今後,BDを対象とした大規模なMWASによって,両疾患におけるエピゲノム要因の類似性や独自性の詳細がさらに明らかとなるであろう.
2)熊本大学大学院生命科学研究部分子脳科学分野
3)東京大学大学院医学系研究科精神医学
4)藤田医科大学医学部精神神経科学
5)湘南鎌倉総合病院精神科
6)東京大学大学院教育学研究科身体教育学
7)理化学研究所脳神経科学研究センター精神疾患動態研究チーム
はじめに
代表的な精神疾患である統合失調症(schizophrenia:SZ)と双極性障害(bipolar disorder:BD)は,共通の臨床症状を有することに加え,遺伝要因と環境要因を共有することが知られており,複雑な遺伝環境相互作用が発症に関与すると考えられている15)17).ゲノムワイド関連解析(genome−wide association study:GWAS)の結果を用いて,さまざまな精神疾患の遺伝要因の重なりを調べた研究では,SZとBDの遺伝要因の重なりが最も大きいことが報告されている6).
エピジェネティクスとは塩基配列の変化を伴わず,ゲノムの有している情報を変化させる機構のことであるが,代表的なエピゲノム要因であるDNAメチル化は,環境要因により変化し,遺伝子発現状態の制御に寄与することから,精神疾患発症にかかわる遺伝環境相互作用において重要な役割を果たすことが考えられる3)16)23).このため,これまでにSZやBDの死後脳や末梢組織を用いたDNAメチル化研究が数多く報告されている5)22)24)30).
近年,SZにおいて,約1,500名の大規模なDNAメチル化関連解析が行われた.この研究では,末梢血から抽出したゲノムDNAから,メチル化シトシン結合蛋白質を利用し高メチル化ゲノムDNA断片を濃縮後,次世代シークエンサーにて網羅的に解析を行っている.その結果,合計100ヵ所以上の有意なDNAメチル化変化が同定された1).上位5ヵ所の候補領域〔family with sequence similarity 63,member B(FAM63B),Rho GTPase activating protein 26(ARHGAP26),CTAGE family member 11,pseudogene(CTAGE11P),TBC1 domain family member 22A(TBC1D22A),染色体16番intergenic領域(IR in chr 16)〕については,独立サンプルを用いたpyrosequencing法で確認しており,いずれも追試されている.この研究は,精神疾患の初の大規模なゲノムワイドメチル化関連解析(methylome―wide association study:MWAS)であり,その後FAM63Bに関してはBDでも同様のメチル化変化が報告されたが27),その他の領域に関してSZとBDのエピゲノム要因の重なりについては検討されていない.
本研究では,遺伝要因と同様に,SZとBDのエピゲノム要因の重なりの程度を調べるために,SZのMWASで同定された上位5ヵ所の候補領域のDNAメチル化解析を,BDの末梢血を用いて行った.
I.研究の方法および結果
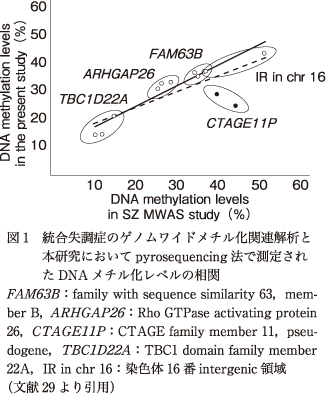
BD患者450名と健常者457名の末梢血由来のDNAを用いてDNAメチル化解析を行った.SZのMWASにおける確認実験に用いられていたpyrosequencing法を用い,解析対象CpG部位,PCRでの増幅領域,pyrosequencingの条件など,なるべく先行研究と同一条件での検討ができるよう努めた.その結果,CTAGE11Pを除くすべての領域のCpG部位におけるDNAメチル化レベルは,先行研究と本研究で高い相関を示した(Pearson's R=0.963,図1).
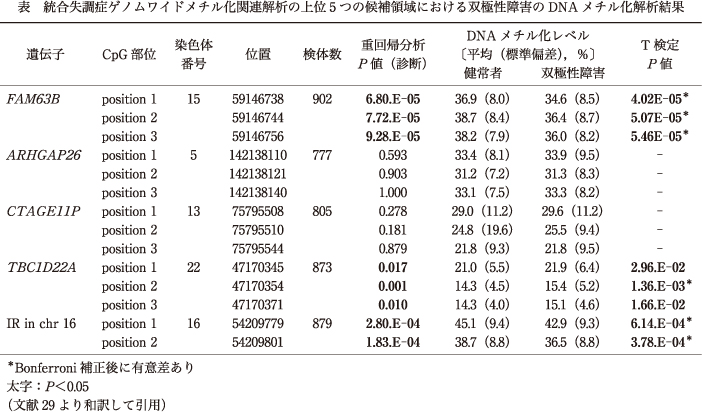
5ヵ所の候補領域におけるDNAメチル化解析の結果,2ヵ所(ARHGAP26,CTAGE11P)の領域におけるCpG部位では,BDと健常者で有意なDNAメチル化レベルの差異は認められなかった.残り3ヵ所のうち2ヵ所(FAM63B,IR in chr 16)の領域におけるCpG部位では,SZと同様に,BDにおいても健常者に比して有意な低メチル化状態が認められた.1ヵ所(TBC1D22A)の領域におけるCpG部位では,SZとは異なり,BDでは健常者に比して有意な高メチル化状態が認められた(表,図2).
これらの領域のDNAメチル化に対する抗精神病薬の影響を検証するため,risperidoneを28日間経口投与したコモンマーモセットの血液を用いた検討を行った.ヒトとマーモセットで進化的に保存されている領域(FAM63B,ARHGAP26,IR in chr 16)(図3a)を検討対象とした.Pyrosequencing法にてDNAメチル化レベルを測定したところ,いずれのCpG部位においても,risperidone投与によるDNAメチル化レベルの変化は認められなかった(図3b).
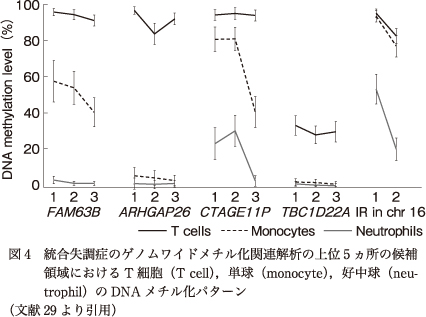
次に,血液細胞種の構成パターンがDNAメチル化に影響している可能性を検討するため,iMETHYLデータベース9)を用いて,各CpG部位におけるT細胞,単球,好中球のDNAメチル化レベルを調べた.結果,T細胞のDNAメチル化レベルは,TBC1D22A以外のほとんどすべてのCpG部位で100%であったのに対し,好中球のDNAメチル化レベルはFAM63B,ARHGAP26,TBC1D22AのCpG部位ではほぼ0%であり,単球のDNAメチル化パターンは領域ごとに異なっていた(図4).
II.考察―本論文の意義,苦労・工夫したことなどを含めて―
本研究では,スウェーデンでのSZのMWASで有意な低メチル化が同定された5つの候補領域について,日本人約900名のBDと健常者の末梢血を用いてDNAメチル化解析を行った.研究者間でのバラつきの影響を除外するために,SZのMWASと同様の方法で同候補領域のメチル化解析を行った結果,CTAGE11P以外のすべての領域において,高い相関を認めた.CTAGE11Pはpseudogeneであり,機能は不明であるが,いくつかの遺伝子のDNAメチル化レベルは人種差が報告されていることから4)18)19),CTAGE11Pにおいて同様の機構が働いている可能性がある.
本研究では,FAM63BとIR in chr 16のCpG部位において,BDで有意な低メチル化が認められたことから,これらのCpG部位におけるDNAメチル化変化はSZとBDで共通である可能性が示唆される.FAM63Bに関しては,MassARRAYを用いたDNAメチル化解析が他グループにより行われており,やはりBDにおいて有意な低メチル化が報告されている27).SZやBDの病態生理におけるFAM63Bの関与は明らかではないが,過去の報告では,ドパミン遺伝子の発現や神経分化にかかわるmicro RNAに調節されるネットワークの一部とされており12)13)26),体内時計の役割を担う松果体に発現量が高いことから20),FAM63BのDNAメチル化変化が精神疾患の病態に関与している可能性が示唆される.
一方で,TBC1D22AのCpG部位に関しては,SZのMWASでは有意な低メチル化が認められていたのに対し,BDでは有意な高メチル化が認められた.TBC1D22Aのメチル化変化は両疾患特有のものである可能性が考えられるが,多重検定後に有意な高メチル化が認められたCpG部位は1ヵ所のみであったため,別のサンプルセットで確認が必要である.
BDで有意なDNAメチル化変化が認められた領域のいずれにおいても,BDと健常者間のDNAメチル化差異はごくわずかなもの(0.8~2.3%)であった.可能な限り,交絡因子の影響を考慮して解析を行ってはいるが,疾患の異種性,加齢,性差,血液細胞の異種性を含むさまざまな要因がDNAメチル化変化に関与している可能性がある.
過去の報告では,抗精神病薬や気分安定薬は脳や末梢血におけるエピゲノム状態に影響を及ぼすことが示唆されている2)7)8)11)21)28).本研究で用いたサンプルの服薬状況は不明であることから,候補領域のDNAメチル化に対する抗精神病薬の影響を調べるため,霊長類であるコモンマーモセットにrisperidoneを投与し,末梢血におけるDNAメチル化変化を調べた.進化的に保存されていた3つの候補領域におけるCpG部位において,risperidoneによるDNAメチル化への影響は認められなかった.残り2つの候補領域については検討できていないが,SZのMWASの所見の大部分は抗精神病薬の影響を受けたものではない可能性が示唆される.
血液はDNAメチル化プロファイルの異なるさまざまな細胞が混在していることから,細胞構成は交絡因子となりうる10)14)25).アレイによるDNAメチル化解析のデータベースを用いた過去の報告では,FAM63Bの1つのCpG部位の近傍のプローブにおいて細胞種によるDNAメチル化差異が検出されている27).本研究では,whole―genome bisulfite sequencingで構築されたデータベースを用いて解析を行った結果,候補領域におけるすべてのCpG部位において細胞種依存性のDNAメチル化パターンが認められた(図4).この結果から,DNAメチル化変化はある程度細胞構成の影響を受けている可能性はあると考えられた.しかし,同じような細胞種依存性のDNAメチル化パターンが認められたCpG部位において,低メチル化と高メチル化の両方が同定されていることから,単純に細胞構成のみでDNAメチル化変化を説明するのは困難であると考えられた.
本研究では,交絡因子となりうる対象患者の重症度や血液・生理検査所見,服薬内容といった臨床情報がなく,マーモセットを用いた投薬実験では,risperidone以外の抗精神病薬や気分安定薬のDNAメチル化に対する影響を調べていないこと,さらに,用いたマーモセットサンプルは少数(N=6)であることから,risperidoneのDNAメチル化に対する影響が偽陰性になっている可能性がある.
おわりに―今後の課題および方向性―
本研究により,遺伝要因に加えて,エピゲノム要因についてもSZとBDで共通している可能性が示唆された.エピゲノム変化は,遺伝子発現制御に直接影響を与えるメカニズムであり,病態に深く関与している可能性が考えられる.一方でGWAS同様に,MWASで同定されるエピゲノム差異も,オッズ比が小さく多因子性(polyepigenetic)の性質を示すことが想定される.今後は,BDを対象としたMWASのような大規模なエピゲノム解析によって,両疾患におけるepigenetic architectureの詳細が明らかとなるであろう.
本論文はPCN誌に掲載された最新の研究論文29)を編集委員会の依頼により,著者の1人が日本語で書き改め,その意義と展望などにつき加筆したものである.
本研究は,熊本大学,東京大学,藤田医科大学,理化学研究所の共同研究である.
なお,本論文に関連して開示すべき利益相反はない.
1) Aberg, K. A., McClay, J. L., Nerella, S., et al.: Methylome-wide association study of schizophrenia: identifying blood biomarker signatures of environmental insults. JAMA Psychiatry, 71; 255-264, 2014![]()
2) Asai, T., Bundo, M., Sugawara, H., et al.: Effect of mood stabilizers on DNA methylation in human neuroblastoma cells. Int J Neuropsychopharmacol, 16 (10); 2285-2294, 2013![]()
3) Bird, A.: DNA methylation patterns and epigenetic memory. Genes Dev, 16 (1); 6-21, 2002![]()
4) Chambers, J. C., Loh, M., Lehne, B., et al.: Epigenome-wide association of DNA methylation markers in peripheral blood from Indian Asians and Europeans with incident type 2 diabetes: a nested case-control study. Lancet Diabetes Endocrinol, 3 (7); 526-534, 2015![]()
5) Connor, C. M., Akbarian, S.: DNA methylation changes in schizophrenia and bipolar disorder. Epigenetics, 3 (2); 55-58, 2008![]()
6) Cross-Disorder Group of the Psychiatric Genomics Consortium: Identification of risk loci with shared effects on five major psychiatric disorders: a genome-wide analysis. Lancet, 381 (9875); 1371-1379, 2013![]()
7) Dong, E., Nelson, M., Grayson, D. R., et al.: Clozapine and sulpiride but not haloperidol or olanzapine activate brain DNA demethylation. Proc Natl Acad Sci U S A, 105 (36); 13614-13619, 2008![]()
8) Dong, E., Chen, Y., Gavin, D. P., et al.: Valproate induces DNA demethylation in nuclear extracts from adult mouse brain. Epigenetics, 5 (8); 730-735, 2010![]()
9) Hachiya, T., Furukawa, R., Shiwa, Y., et al.: Genome-wide identification of inter-individually variable DNA methylation sites improves the efficacy of epigenetic association studies. NPJ Genom Med, 2; 11, 2017![]()
10) Houseman, E. A., Accomando, W. P., Koestler, D. C., et al.: DNA methylation arrays as surrogate measures of cell mixture distribution. BMC Bioinformatics, 13; 86, 2012![]()
11) Houtepen, L. C., van Bergen, A. H., Vinkers, C. H., et al.: DNA methylation signatures of mood stabilizers and antipsychotics in bipolar disorder. Epigenomics, 8 (2); 197-208, 2016![]()
12) Huang, T., Liu, Y., Huang, M., et al.: Wnt1-cre-mediated conditional loss of Dicer results in malformation of the midbrain and cerebellum and failure of neural crest and dopaminergic differentiation in mice. J Mol Cell Biol, 2 (3); 152-163, 2010![]()
13) Huang, W., Li, M. D.: Differential allelic expression of dopamine D1 receptor gene (DRD1) is modulated by microRNA miR-504. Biol Psychiatry, 65 (8); 702-705, 2009![]()
14) Jaffe, A. E., Irizarry, R. A.: Accounting for cellular heterogeneity is critical in epigenome-wide association studies. Genome Biol, 15 (2); R31, 2014![]()
15) Kato, T.: Whole genome/exome sequencing in mood and psychotic disorders. Psychiatry Clin Neurosci, 69 (2); 65-76, 2015![]()
16) Labrie, V., Pai, S., Petronis, A.: Epigenetics of major psychosis: progress, problems and perspectives. Trends Genet, 28 (9); 427-435, 2012![]()
17) Lichtenstein, P., Yip, B. H., Björk, C., et al.: Common genetic determinants of schizophrenia and bipolar disorder in Swedish families: a population-based study. Lancet, 373 (9659); 234-239, 2009![]()
18) Luo, M., Zhou, X., Ji, H., et al.: Population difference in the associations of KLOTH promoter methylation with mild cognitive impairment in Xinjiang Uygur and Han populations. PLoS One, 10 (7); e0132156, 2015![]()
19) Ma, W., Zhou, X., Ji, H., et al.: Population difference in the association of BDNF promoter methylation with mild cognitive impairment in the Xinjiang Uygur and Han populations. Psychiatry Res, 229 (3); 926-932, 2015![]()
20) Macchi, M. M., Bruce, J. N.: Human pineal physiology and functional significance of melatonin. Front Neuro-endocrinol, 25 (3-4); 177-195, 2004![]()
21) Melas, P. A., Rogdaki, M., Ösby, U., et al.: Epigenetic aberrations in leukocytes of patients with schizophrenia:association of global DNA methylation with antipsychotic drug treatment and disease onset. FASEB J, 26 (6); 2712-2718, 2012![]()
22) Nishioka, M., Bundo, M., Kasai, K., et al.: DNA methylation in schizophrenia: progress and challenges of epigenetic studies. Genome Med, 4 (12); 96, 2012![]()
23) Petronis, A.: Epigenetics as a unifying principle in the aetiology of complex traits and diseases. Nature, 465 (7299); 721-727, 2010![]()
24) Pidsley, R., Mill, J.: Epigenetic studies of psychosis: current findings, methodological approaches, and implications for postmortem research. Biol Psychiatry, 69 (2); 146-156, 2011![]()
25) Reinius, L. E., Acevedo, N., Joerink, M., et al.: Differential DNA methylation in purified human blood cells: implications for cell lineage and studies on disease susceptibility. PLoS One, 7 (7); e41361, 2012![]()
26) Sempere, L. F., Freemantle, S., Pitha-Rowe, I., et al.: Expression profiling of mammalian microRNAs uncovers a subset of brain-expressed microRNAs with possible roles in murine and human neuronal differentiation. Genome Biol, 5 (3); R13, 2004![]()
27) Starnawska, A., Demontis, D., McQuillin, A., et al.: Hypomethylation of FAM63B in bipolar disorder patients. Clin Epigenetics, 8; 52, 2016![]()
28) Sugawara, H., Bundo, M., Asai, T., et al.: Effects of quetiapine on DNA methylation in neuroblastoma cells. Prog Neuropsychopharmacol Biol Psychiatry, 56; 117-121, 2015![]()
29) Sugawara, H., Murata, Y., Ikegame, T., et al.: DNA methylation analyses of the candidate genes identified by a methylome-wide association study revealed common epigenetic alterations in schizophrenia and bipolar disorder. Psychiatry Clin Neurosci, 72 (4); 245-254, 2018![]()
30) Teroganova, N., Girshkin, L., Suter, C. M., et al.: DNA methylation in peripheral tissue of schizophrenia and bipolar disorder: a systematic review. BMC Genet, 17; 27, 2016![]()